raw_data <-
"Deutschland ist ein Land mit 83,3 Mln. Einwohnern und sein Hauptstadt
ist Berlin; Russland hat 144,1 Mln. Einwohner, Hauptstadt ist Moskau;
Weiteres Land ist Usbekistan mit 35,5 Mln. Einwohnern, sein Hauptstadt
ist Taschkent. Letztes Land sind USA mit 341,2 Mln. Einwohnern, die
Hauptstadt ist Washington, D.C."
Set Up
Der Codeblock bzw. Input wird in R hellcyan und in Python gelb dargestellt. Zeilenanfang im Output ist mit #> belegt.
Code
cat("Hallo R Lover!")
#> Hallo R Lover!Code
print("Hallo Python Lover!")
#> Hallo Python Lover!Eine Zusatzinfo ist im Boxe wie unten geschrieben kann duerch ausklappen angezeigt werden:
Data Structures
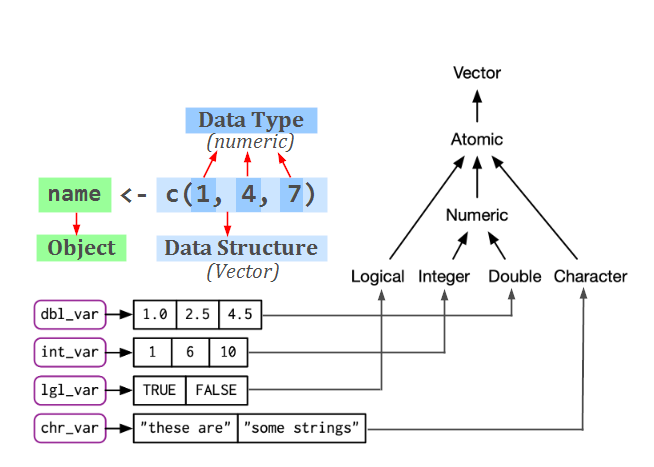
Es gibt einmal die Basic Data Types, die bei der Zuweisung eines Werts oder bei der Erzeugung einer Variable bzw. eines Objekts festlegen, welche Werte dieses Objekt/Variable annehmen kann und welche Operationen auf diese Objekte anzuwenden sind. Z.B. eine Zahl 42 der Variable namens beispielsweise var_zahl zugewiesen und mit dem Typ Integer gespeichert werden. Dann kan weitere Berechnugen mit dieser Variable wie Multioplikation, Addition usw. durchgeführt werden. So können Daten als systematisch kodierte Informationen über die reale Welt definiert (Weidmann 2023) werden. Daten sind für uns dann vom Nutzen, wenn sie in einer Format sind, so dass sie gespeichert, geteilt, analisiert und auf sie irgendwelche Operationen und Fubktionen angewendet werden können. Das bestimmt die Data Dstructure, also Oraganisation und Speicherung von Daten. Was gemeint ist, möchte ich durch ein Beispiel in Anlehnung an (Weidmann 2023) zeigen.
Man kann das Object raw_data im Grunde als eine Datenbank sehen, welche die Informationen über vier Länder, ihre Hauptstädte und ihre Bevölkerungszahl enthält. Für Menschen ist einfach, aus diesem Text die Informationen zu extrahieren, wie z.B. welches Land hat größere Population, welche Population ist die größte oder kleinste usw. Aber für Komputer ist nicht verständlich, sie rechnerisch zu verarbeiten, weil die Daten keine Struktur haben. Wir könnten aber diese Daten mit dem gleichen Inhalt anders speichern, also mit einer bestimmten Struktur z. B. als datafarame. Somit sind Daten dem Komputer verständlich, so dass er rechnerisch verarbeiten kann.
show the code
#> country population capital
#> 1 Germany 83.3 Berlin
#> 2 Uzbekistan 35.5 Tashkent
#> 3 Russia 144.1 Moscow
#> 4 USA 341.2 WashingtonUm die Daten zu speichern, wurde im zweiten Beispiel data.frame von R verwendet, eine Data Structure von R für Tabellen, wie es in Python dafür pandas gibt. Jedes Land hat verschieden Typen von Informationen (county ist character also string, population ist double also float). Im Verglich zu den im ersten Beispiel als normaler Text (raw_data) gespeicherten Daten sind als dataframe bzw Tabelle gespeicherte (df_r) Daten leichter zu verstehen und zu verarbeiten.
show the code
#> Sum of population: 604.1
#> Max of Population: 341.2
#>
#> Summary of data:
#> country population capital
#> Length:4 Min. : 35.50 Length:4
#> Class :character 1st Qu.: 71.35 Class :character
#> Mode :character Median :113.70 Mode :character
#> Mean :151.03
#> 3rd Qu.:193.38
#> Max. :341.20show the code
#> Sum of population: 604.0999999999999
#> Sum of population: 341.2
#>
#> Summary of data:
#> population
#> count 4.000000
#> mean 151.025000
#> std 134.346799
#> min 35.500000
#> 25% 71.350000
#> 50% 113.700000
#> 75% 193.375000
#> max 341.200000How is the data are structured in R and Python
Egal, ob wir die Daten in R oder in Python bearbeiten, in beiden Sprachen werden sie als Object gespeichert. Objekte können erstellt werden, indem man z.B einer Variable (Name des Objekts) den Wert (Objekt) zuweist. Sagen wir, wir erzeugen ein Objekt namens age und weiesen ihm den Wert 35 (Alter): age <- 35 (in R) und age = 35 (in Python). Aber meistens will man nicht manulell Tausende von Objekten (age1, age2, age3, …) erstellen, sondern eine Liste namens ages haben, die alle Alter (Objekte) enthält. Daraus wird eine gruppierte Datenstruktur, aslo ein Object, das aus mehreren Einzelobjecten zusammengesetzt sind. Davon gibt es unterschiedliche, wie Lists, Vectors, Arrays, Series, Dictionaries etc. Sie unterscheiden sich durch ihre Format, Struktur, Methoden usw., aber Kernpunkt ist gleich: Es werden mehrere Objekte in ein gruppiertes bzw kombiniertes Objekt (in einer von dieser Datenstruktur) gespeichert.
show the code
#> [1] 35 24 42 24 52 35 37
#> [1] "numeric"
#> [1] "Ali" "Temur" "Ben" "Brandon" "John" "Doe" "Dirk"
#> [1] "character"c() in R
In R werden Sequenzen oder Reihen mit c() (c wie combine) erzeugt, das ist wie man in Python die Objekte bzw. Daten in Form [] eingibt.
show the code
#> [35, 24, 42, 24, 52, 35, 37]
#> <class 'list'>
#> ['Ali', 'Temur', 'Ben', 'Brandon', 'John', 'Doe', 'Dirk']
#> <class 'list'>Sie sind in Python und R ähnlich, nur Obekt in R heiß Vektor und in Python heiß Liste. Es gibt auch andere Datenstrukturen in Python und R, die nach Eigenschaften identisch sind oder sich unterscheiden. Beim Vergleich die Datenstrukturen in R und Python, denke ich, ist es aber wichtig, dass die Objekte homogen oder heterogen sind.
Data Structures in R
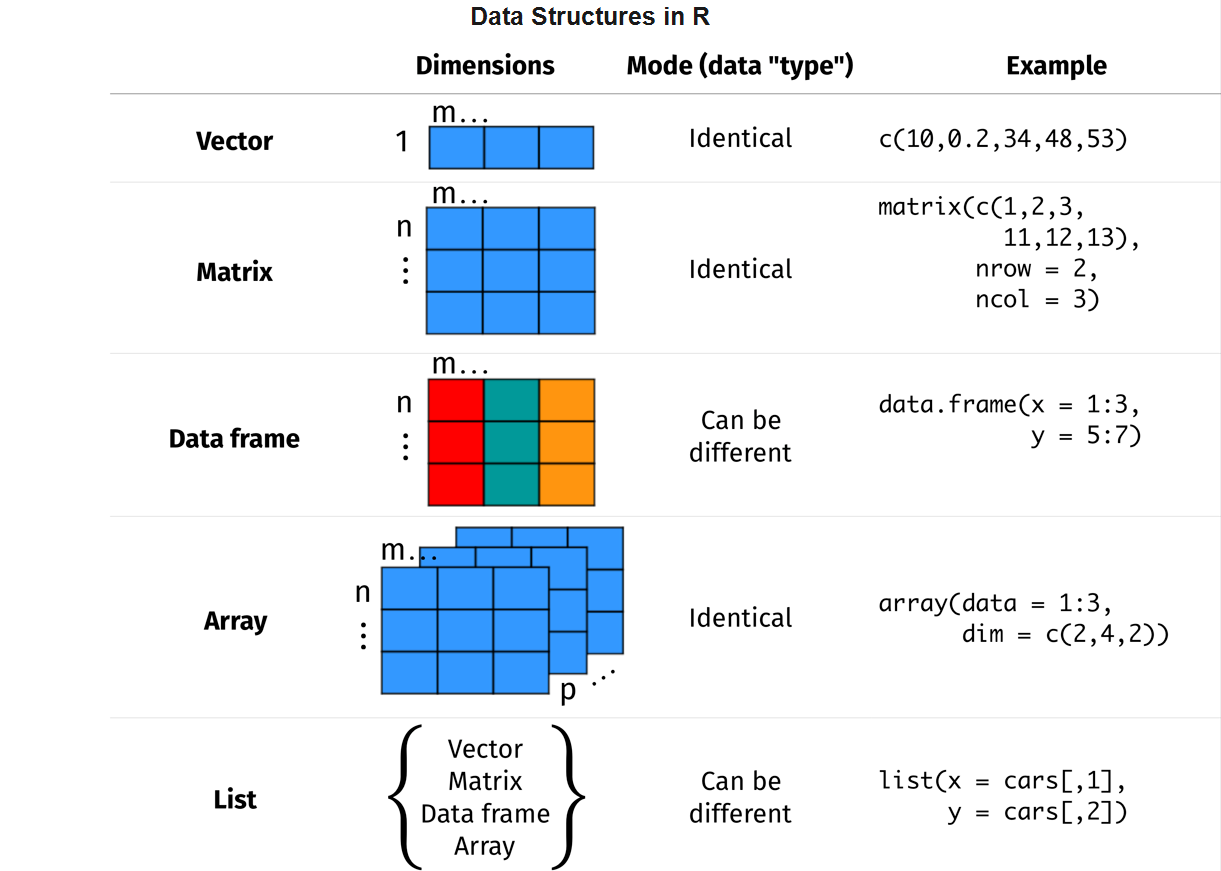
Wichtige Datenstrukturen in R könen dadurch, wie viele Dimensionen ein Objekt hat - ein demensional (Vektor), zwei demensional (Matrix) und mehr (Arrays) und dadurch, ob die Elemente eines Objekts homogen - alle Elemente von gleicher Art z.B. nur Text, Zahlen sind, oder ob die Elemente heterogen - alle Elemente unterschiedlich sind, gekenzeichnet werden. Anhand dieser Merkmalen kann man die zentralen Objektarten wie folgt zusammenfassen (Sauer 2019):
| Dimension | Homogen | Heterogen |
|---|---|---|
| 1d | Vector | List |
| 2d | Matrix | Dataframe |
| nd | Array |
Vektoren sind eine der wichtigsten Datensrtukturen in R und stellen ein zentrales Elemnt in R-Objekten dar. Es gibt vier Arten von Atomar- bzw. Reinvektoren: Logical, Integer, Double und Character (welche stings entält), wobei Integer und Double sich zum Datentyp Numeric zusammenfassen lassen. Hier eine Übersicht von atomaren Vektoren nach (Wickham 2019).
Data Structute in Python
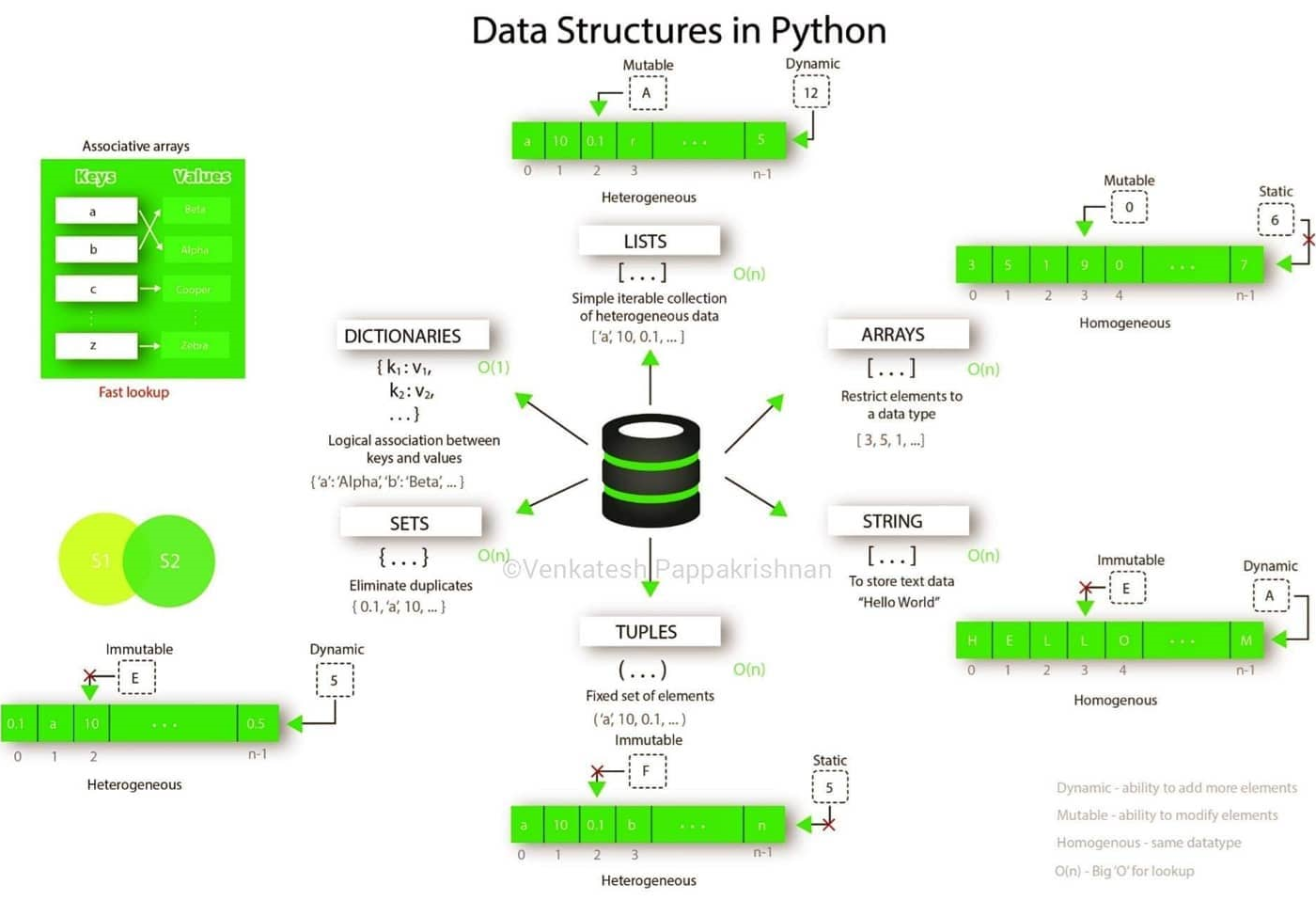
Datenstrukturen können also als „Container“ betrachtet werden, die Daten nach Typ organisieren und gruppieren. Die sogenannten wichtigen Built-in Data Structures in Python sind Liste (list), Menge (set), Dictionaries (dict) und Tupel (tuple). Jeder Datentypen haben eigene Struktur und Eigenschaften
- lists
- ditionaries
- tuples
- sets
Darüber hinaus gibt Arrays und Dataframes(Tabellen):
- arrays von NumPy (Numerical Python)
- DataFrame von Pandas
Im Folgenden sind die Data Structures in R und Python grafisch dargestellt.
Homogeneous data structure
Wie oben schon erwähnt, homogene Datenstrukturen enthalten die Daten (Objekte oder Elemente) desselben Typs. Meistents werden die Daten auf dieser Art gespeichert. Dazu gehören in R Vektoren (1d-NumPy-Arrays in Python), Matizien (2d-NumPy-Arrays in Python) und Arrays (nd-NumPy-Arrays in Python).
vector in R and arrays & Series in Python
Vektoren in R sind eindimensional und homogen. Mit dieser Eigenschaft haben sie Gemeinsamkeit mit 1d-NumPy-Arrays und Pandas Series in Python. Vektoren in R sind auch ähnlich wie die Liste in Python, aber nur dann, wenn die Daten in der Liste von Python den gleichen Typ haben.
Creating
– with c() (like [] in Python)
show the code
#> [1] 1 7 2 4 5 8 2 9
#> [1] "numeric"
#> [1] "a" "b" "c" "z" "f"
#> [1] "character"
#> [1] FALSE FALSE FALSE FALSE FALSE
#> [1] "logical"
#> [1] 0 0 0 0 0 0 0
#> [1] "numeric"– with seq(from, to, by) (like range(from, to, by) in Python or arange() from NumPy)
show the code
#> [1] 2 3 4 5 6 7 8 9
#> [1] 1 2 3 4 5
#> [1] 3 6 9
#> [1] 3.00 4.75 6.50 8.25 10.00
#> [1] 11.0 12.5 14.0 15.5 17.0– with rep() (like np.repeat() from NumPy or pd.rep() from Pandas)
– with sample() or runif() (like random.choice() from NumPy)
show the code
smp1 <- sample(seq(10), size = 5, replace = FALSE); smp1
vec <- c("negative","neutral", "positive")
smp2 <- sample(vec, size = 5, replace = TRUE); smp2
# vector with 5 elements:
run1 <- runif(5); run1
run2 <- runif(3, min = 1, max = 10); run2
run3 <- floor(runif(15, min=1, max=100)); run3
# normal distribution
rn <- rnorm(5); rn#> [1] 1 8 10 6 9
#> [1] "neutral" "positive" "positive" "negative" "negative"
#> [1] 0.2332719 0.4469084 0.9075884 0.2411691 0.6932269
#> [1] 2.734668 4.615119 9.273173
#> [1] 75 80 22 37 76 89 47 36 21 49 25 19 73 46 40
#> [1] -0.6840332 0.7098267 -0.8828714 -0.5181750 0.2727017– with [] like c() in R
show the code
#> [1, 7, 2, 4, 5, 8, 2, 9]
#> <class 'list'>
#> [False False False False False]
#> <class 'numpy.ndarray'>
#> [0 0 0 0 0 0 0]
#> <class 'numpy.ndarray'>
#> [1 1 1 1 1 1 1]
#> <class 'numpy.ndarray'>– with arange() like seq() in R
show the code
#> [0, 1, 2, 3, 4]
#> [2, 3, 4, 5, 6, 7, 8, 9]
#> array([2, 3, 4, 5, 6, 7, 8, 9])
#> array([3, 6, 9])
#> (array([ 3. , 4.75, 6.5 , 8.25, 10. ]), 1.75)– with np.repeat() or pd.Series.repeat() like rep() in R
show the code
#> [1, 2, 3, 1, 2, 3, 1, 2, 3]
#> array([1, 2, 3, 1, 2, 3, 1, 2, 3])
#> array([1, 1, 1, 2, 2, 2, 3, 3, 3])
#> 0 1
#> 0 1
#> 1 2
#> 1 2
#> 2 3
#> 2 3
#> dtype: int64– with random() like sample() or runif() in R
show the code
from numpy import random
lis = ["negative","neutral", "positive"]
rd1 = random.choice(lis, size=5, replace = True); print(rd1)
rd2 = random.rand(5); rd2 # 5 random numbers
rd3 = random.randint(100, size=(5)); rd3 # integer random numbers from 0 to 100
rd4 = random.randn(4); rd4 # standard normal distribution#> ['neutral' 'neutral' 'neutral' 'neutral' 'negative']
#> array([0.37621427, 0.340763 , 0.43995926, 0.61406061, 0.32292481])
#> array([32, 26, 10, 59, 86])
#> array([ 0.47682804, 0.89101471, -0.21079475, -0.00142282])Slicing
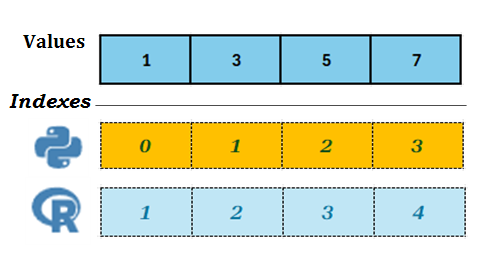
Eine der wichtigsten nützlichsten Operationen, die man auf Vektoren, Listen und eindimensionale Arrays anwenden kann, ist Indexierung. Dabei kann man einzelnes Element oder Teilmenge von Elementen durch ihre Position (Index) auswählen.
# In Python
lst = [1,3,5,7]
lst[0] # first element
lst[1] # second element
lst[-1] # last element
lst[::-1] # backward# In R
# Used Variable lst created in Python Chunk
py$lst[1] # first element
py$lst[2] # second element
py$lst[[length(py$lst)]] # last element
rev(py$lst) # backward#> 1
#> 3
#> 7
#> [7, 5, 3, 1]#> [1] 1
#> [1] 3
#> [1] 7
#> [1] 7 5 3 1Hier noch weitere Beispiele für Teilmengen und logisches Indexieren. Bei der logischen Indexierung werden die Elemente zurückgegeben, deren logischer Wert gleich TRUE ist.
show the code
age <- c(18, 26, 32, 41, 55, 22, 35, 37, 20, 25)
age[1:5] # from first until fifth element
age[3:7] # from third until seventh element
age[5:length(age)] # from fifth until last element
age[c(2,5,7)] # second, fifth and seventh element
age < 30 # logic
age[age < 30] # age under 30
age[which(age < 30)] # with which()#> [1] 18 26 32 41 55
#> [1] 32 41 55 22 35
#> [1] 55 22 35 37 20 25
#> [1] 26 55 35
#> [1] TRUE TRUE FALSE FALSE FALSE TRUE FALSE FALSE TRUE TRUE
#> [1] 18 26 22 20 25
#> [1] 18 26 22 20 25Elemente können auch beim Namen angesprochen werden, aber nur dann, wenn sie benannt sind. Das könnte mann auch in Series von Pandas machen, in dem man Element durch den Indexnamen auswählt:
show the code
age = np.array(r.age) # create array from r vector age
age[:5] # from first until fifth element
age[2:7] # from third until seventh element
age[4:] # from fifth until last element
[age[2], age[5], age[7]] # second, fifth and seventh elem
age < 30 # logic
age[age < 30] # age under 30
age[np.where(age<30)] # with np.where()#> array([18., 26., 32., 41., 55.])
#> array([32., 41., 55., 22., 35.])
#> array([55., 22., 35., 37., 20., 25.])
#> [32.0, 22.0, 37.0]
#> array([ True, True, False, False, False, True, False, False, True,
#> True])
#> array([18., 26., 22., 20., 25.])
#> array([18., 26., 22., 20., 25.])Auch bei Series kann man Element oder Elemente durch ihre Indexen oder Indexnamen ansprechen, das geht wie bei named vectors in R
show the code
#> bad 2
#> good 3
#> medium 4
#> best 5
#> dtype: int64
#> 3
#> best 5
#> bad 2
#> dtype: int64
#> 4
#> good 3
#> medium 4
#> best 5
#> dtype: int64matrix in R and 2d-Array in Python
Matrizen in R sind zweidimensionale Vektoren, deren Elemente numerisch sind, also homogen. Sie werden durch die Funktion matrix(data, nrow, ncol) erstellt. In Python gibt es keine Funktion explizit für die Erstellung von Matrizen. Matrizen in Python sind im Prinzip 2d-NumPy-Arrays. Für die Slicing kommen hier auch gleiche Methoden wie bei Vektoren oder 1d-NumPy-Arrys (vorheriges Kapitel) zur Anwenung, eben werden neben Rowindex auch Columnindex in rechteckigen Klammern [] eingegeben.
show the code
matrix_c <- matrix(seq(12), nrow = 3, ncol = 4, byrow = FALSE )
cat("3x4-Matix filling by columns (matrix_c):\n");print(matrix_c)
# Dimension of matrix_c
glue("Dimension of matrix_c: {dim(matrix_c)}")
# Counts of rows & Cols from matrix_c
glue("Shape of matrix_c: ({nrow(matrix_c)}, {ncol(matrix_c)})")
matrix_r <- matrix(seq(12), nrow = 4, ncol = 3, byrow = TRUE )
cat("\n4x3-Matix filling by rows (matrix_r):\n");print(matrix_r)
# Dimension of matrix_r
glue("Dimension of matrix_r: {dim(matrix_r)}")
# Counts of rows & Cols from matrix_r
glue("Shape of matrix_r: ({nrow(matrix_r)}, {ncol(matrix_r)})")
#
cat("\nSlicing:\n")
matrix_c[2,3] # row 2, col 3
matrix_c[,4] # column 4
matrix_c[, c(2, 4)] # columns 2 and 4
matrix_r[2,] # row 2
# Name cols & rows
cat('\nname the columns and rows of matrix "matrix_r":\n')
colnames(matrix_r) <- c("col1", "col2", "col3")
rownames(matrix_r) <- c("row1", "row2", "row3", "row4")
matrix_r#> 3x4-Matix filling by columns (matrix_c):
#> [,1] [,2] [,3] [,4]
#> [1,] 1 4 7 10
#> [2,] 2 5 8 11
#> [3,] 3 6 9 12
#> Dimension of matrix_c: 3
#> Dimension of matrix_c: 4
#> Shape of matrix_c: (3, 4)
#>
#> 4x3-Matix filling by rows (matrix_r):
#> [,1] [,2] [,3]
#> [1,] 1 2 3
#> [2,] 4 5 6
#> [3,] 7 8 9
#> [4,] 10 11 12
#> Dimension of matrix_r: 4
#> Dimension of matrix_r: 3
#> Shape of matrix_r: (4, 3)
#>
#> Slicing:
#> [1] 8
#> [1] 10 11 12
#> [,1] [,2]
#> [1,] 4 10
#> [2,] 5 11
#> [3,] 6 12
#> [1] 4 5 6
#>
#> name the columns and rows of matrix "matrix_r":
#> col1 col2 col3
#> row1 1 2 3
#> row2 4 5 6
#> row3 7 8 9
#> row4 10 11 12show the code
matriks_c = np.arange(1,13).reshape(3,4, order = "F")
print(f"3x4-Matix filling by columns (matriks_c):\n{matriks_c}")
# Dimension of matrix matriks_c
print(f"Dimension of matrix matriks_c: {matriks_c.ndim}")
# Counts of rows & Cols from matriks_c
print(f"Shape of matriks_c: {matriks_c.shape}")
# np.shape(matriks_c)[0]; np.shape(matriks_c)[1]
matriks_r = np.arange(1,13).reshape(4,3, order = "C")
print(f"\n4x3-Matix filling by rows (matriks_r):\n{matriks_r}")
# Dimension of matrix matriks_r
print(f"Dimension of matrix matriks_r: {matriks_r.ndim}")
# Counts of rows & Cols from matriks_r
print(f"Shape of matriks_r: {matriks_r.shape}")
# np.shape(matriks_r)[0]; np.shape(matriks_r)[1]
#
print("\nSlicing:")
matriks_c[1,2] # row 2, col 3
matriks_c[:, 3] # column 4
matriks_c[:, [1,3]] # columns 2 and 4
matriks_r[1, :] # row 2
# Name cols & rows
print('\nname the columns and rows of matrix "matriks_r":')
colnames = ["col1", "col2", "col3"]
rownames = ["row1", "row2", "row3", "row4"]
pd.DataFrame(matriks_r, columns = colnames, index = rownames)#> 3x4-Matix filling by columns (matriks_c):
#> [[ 1 4 7 10]
#> [ 2 5 8 11]
#> [ 3 6 9 12]]
#> Dimension of matrix matriks_c: 2
#> Shape of matriks_c: (3, 4)
#>
#> 4x3-Matix filling by rows (matriks_r):
#> [[ 1 2 3]
#> [ 4 5 6]
#> [ 7 8 9]
#> [10 11 12]]
#> Dimension of matrix matriks_r: 2
#> Shape of matriks_r: (4, 3)
#>
#> Slicing:
#> 8
#> array([10, 11, 12])
#> array([[ 4, 10],
#> [ 5, 11],
#> [ 6, 12]])
#> array([4, 5, 6])
#>
#> name the columns and rows of matrix "matriks_r":
#> col1 col2 col3
#> row1 1 2 3
#> row2 4 5 6
#> row3 7 8 9
#> row4 10 11 12Mehrdiminsionale Arrays
(Mehrdimensionale)Arrays in R werden mit der Funktion array(vector, dim = c(nrow, ncol, nmat)) erstellt. Dabei ist vector Datenreihe vom gleichen Typ, nrow und ncol sind Zeilen- und Spaltennummer, nmat gibt die Dimension an, also die Zahl von nrow*ncol Matrizen im Array.
Für die Erstellung von Arrays in Python gibt es mehrere Funktionen wie numpy.arange(), numpy.zeros(), numpy.ones(). Im Folgenden wird ein 3d-Array in R und Python sowie Slicing und Summenberechnung dargestellt.
Im Folgenden wird ein Beispiel für 3d-Array visuell und Slicing und Summenberechnung bei diesem Array praktisch dargestellt.
Slicing
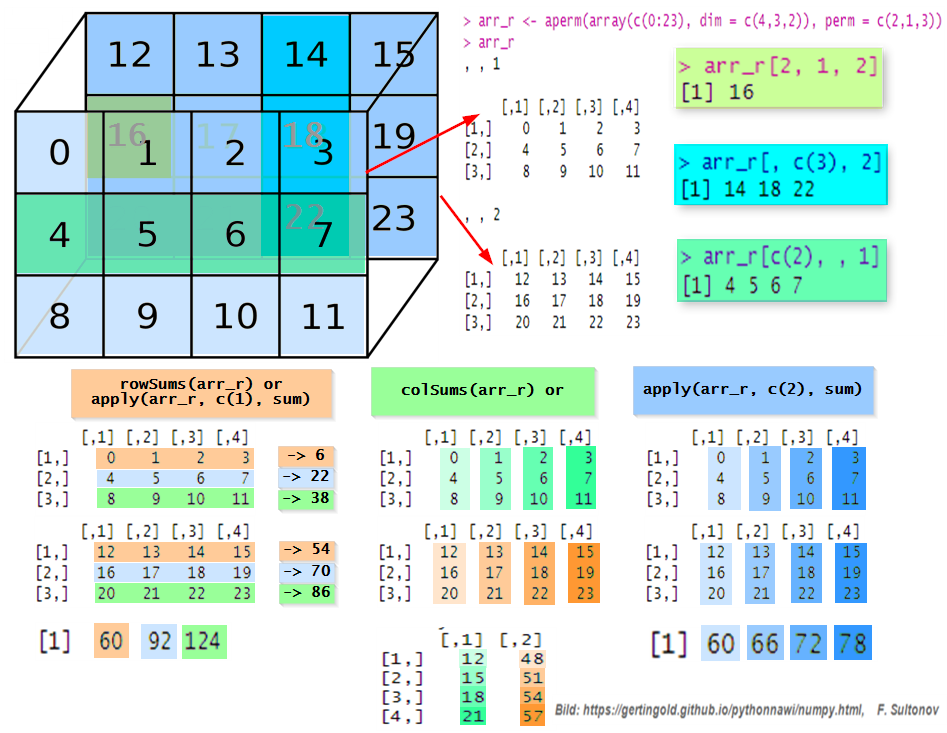
Wie bei Vektoren, oder 1d- oder 2d-Arrays kann auch bei 3d-Arrays ein Element oder eine Teilmenge aus dem Array abgeschnitten werden. Dabei werden Index von dem Element oder Indizies von der Teilmenge in bekannten rechteckigen Klammern [] eingegeben. Im Beispiel hat der Array shape = (3, 4, 2). Das gibt die Funktion in R dim(array) und in Python array.shape zurück. im Beispiel gibt 3 - Zeilen, 4 - Spalten und 2 - Dimension an. D.h., der Array hat 2 Matrizen jeweils mit 3 Zeilen und 4 Spalten. Beim Indexieren muss man dementsprechen in richtige Position eingeben. Z.B. arr_r[, , 2] gibt den zweiten Matrix mit allen Zeilen und Spalten zurück.
show the code
# Create 3d-array
arr_r <- array(c(0:23), dim = c(4,3,2))
arr_r <- aperm(arr_r, perm = c(2,1,3)); arr_r
cat("Shape: ",dim(arr_r), "\n")
cat("\n1st matrix/2d-Array of the 3d-Array (arr_r):\n")
arr_r[, , 1]
cat("\nElement in 2nd row and 1st col of the 2nd matrix from 3d-Array (arr_r):\n")
arr_r[2, 1, 2]
cat("\n3rd column elements of the 2nd matrix from 3d-Array (arr_r):\n")
arr_r[, c(3), 2]
cat("\n2nd row elements of the 1st matrix from 3d-Array (arr_r):\n")
arr_r[c(2), , 1]
cat("\nCheck if elemnt (9) exist in the array:\n")
9 %in% arr_r#> , , 1
#>
#> [,1] [,2] [,3] [,4]
#> [1,] 0 1 2 3
#> [2,] 4 5 6 7
#> [3,] 8 9 10 11
#>
#> , , 2
#>
#> [,1] [,2] [,3] [,4]
#> [1,] 12 13 14 15
#> [2,] 16 17 18 19
#> [3,] 20 21 22 23
#>
#> Shape: 3 4 2
#>
#> 1st matrix/2d-Array of the 3d-Array (arr_r):
#> [,1] [,2] [,3] [,4]
#> [1,] 0 1 2 3
#> [2,] 4 5 6 7
#> [3,] 8 9 10 11
#>
#> Element in 2nd row and 1st col of the 2nd matrix from 3d-Array (arr_r):
#> [1] 16
#>
#> 3rd column elements of the 2nd matrix from 3d-Array (arr_r):
#> [1] 14 18 22
#>
#> 2nd row elements of the 1st matrix from 3d-Array (arr_r):
#> [1] 4 5 6 7
#>
#> Check if elemnt (9) exist in the array:
#> [1] TRUEOperationen
Um die Summe bei Arrays in R zu berechnen, kann man die Funktion rowSums()- Summe von Zeilen, colSums()- Summe von Spalten benutzen, es gibt auch in R die Funktion sum() wie in Python, diese kann man hier mit der Funktion apply benutzen.
show the code
#> , , 1
#>
#> [,1] [,2] [,3] [,4]
#> [1,] 0 1 2 3
#> [2,] 4 5 6 7
#> [3,] 8 9 10 11
#>
#> , , 2
#>
#> [,1] [,2] [,3] [,4]
#> [1,] 12 13 14 15
#> [2,] 16 17 18 19
#> [3,] 20 21 22 23
#>
#> Sum by row-wise with rowSums:
#> [1] 60 92 124
#>
#> Sum by row-wise with apply, same like rowSums:
#> [1] 60 92 124
#>
#> Sum by column-wise with colSums:
#> [,1] [,2]
#> [1,] 12 48
#> [2,] 15 51
#> [3,] 18 54
#> [4,] 21 57
#>
#> Sum by column-wise with apply
#> [1] 60 66 72 78Slicing
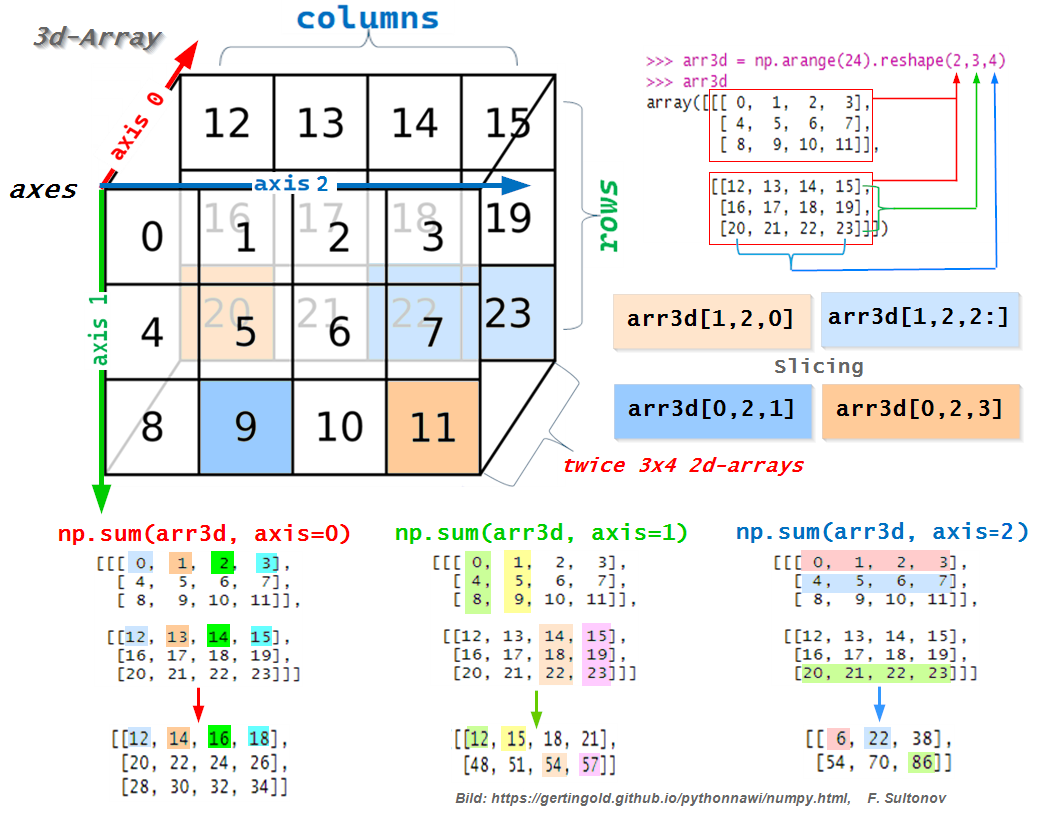
Die Funktion array.shape geibt shape = (2, 3, 4) von dem Array im Beispiel oben im Bild zurück. D.h., als erstes die Dimension - 2, als zweites Zeilen - 3 und als viertes Spalten - 4 zurückgegeben. D.h., der Array hat 2 Matrizen jeweils mit 3 Zeilen und 4 Spalten. Man möge sich einen Zauberwürfel vorstellen. Wenn man diesen in der Mitte in Scheiben schneidet und in zwei teilt, wäre jeder Teil ein Matrix oder 2d-Array. Man könnte auch diese Teile als Schicht (layer/sheet) vorstellen. Beim Indexieren muss man dementsprechen in richtige Position eingeben. Z.B. arr_r[1, :, :] gibt den zweiten Matrix mit allen Zeilen und Spalten zurück.
In R wird die Reihenfolge in Shape anders dargestellt als in Python für gleichen Array, shape in R = (3, 4, 2), shape in Python = (2, 3, 4). In R werden erst Zeilen, dann Spalten und als letztes Dimension) dargestellt. In Python kommt erst Dimension, dann Zeilen und Spalten.
show the code
# create 3d-Array
arr3d = np.arange(24).reshape(2,3,4);arr3d
print(f"\nShape: {arr3d.shape}")
print(f"\n1st 2d-Array (sheet,matrix, layer) of the 3d-Array (arr3d):")
arr3d[0, :, :] # or arrd[0]
print(f"\n3rd column elements of the 2nd sheet from 3d-Array (arr3d):")
arr3d[1, :, 2]
print(f"\n2nd row elements of the 1st sheet from 3d-Array (arr3d):")
arr3d[0, 1, :]
print(f"\nElement (20) in 3rd row and 1st col of the 2nd sheet from 3d-Array (arr3d):")
arr3d[1, 2, 0]
print(f"\nElements (22, 23) from 3rd row and columns 3 and 4 in the 2nd sheet:")
arr3d[0, 2, 2:]
print(f"\nElement (9) in 3rd row and 2nd col of the 1st sheet from 3d-Array (arr3d):")
arr3d[0, 2, 1]
print(f"\nElement (11) in 3rd row and last col of the 1st sheet from 3d-Array (arr3d):")
arr3d[0, 2, 3]#> array([[[ 0, 1, 2, 3],
#> [ 4, 5, 6, 7],
#> [ 8, 9, 10, 11]],
#>
#> [[12, 13, 14, 15],
#> [16, 17, 18, 19],
#> [20, 21, 22, 23]]])
#>
#> Shape: (2, 3, 4)
#>
#> 1st 2d-Array (sheet,matrix, layer) of the 3d-Array (arr3d):
#> array([[ 0, 1, 2, 3],
#> [ 4, 5, 6, 7],
#> [ 8, 9, 10, 11]])
#>
#> 3rd column elements of the 2nd sheet from 3d-Array (arr3d):
#> array([14, 18, 22])
#>
#> 2nd row elements of the 1st sheet from 3d-Array (arr3d):
#> array([4, 5, 6, 7])
#>
#> Element (20) in 3rd row and 1st col of the 2nd sheet from 3d-Array (arr3d):
#> 20
#>
#> Elements (22, 23) from 3rd row and columns 3 and 4 in the 2nd sheet:
#> array([10, 11])
#>
#> Element (9) in 3rd row and 2nd col of the 1st sheet from 3d-Array (arr3d):
#> 9
#>
#> Element (11) in 3rd row and last col of the 1st sheet from 3d-Array (arr3d):
#> 11Operationen
Um die Summe bei Arrays in Paython zu berechnen, muss axis eingegeben werden, je nachdem, auf welche Weise man die Summe berechnen will. Gibt man 0 ein, wird die Summe über n-Matrizen gebildet. Bei 1 wird die Summe über spaltenweise und bei 2 zeilenweise berechnet.
show the code
#> a 3d-array - arr3d:
#> array([[[ 0, 1, 2, 3],
#> [ 4, 5, 6, 7],
#> [ 8, 9, 10, 11]],
#>
#> [[12, 13, 14, 15],
#> [16, 17, 18, 19],
#> [20, 21, 22, 23]]])
#>
#> Shape:
#> (2, 3, 4)
#>
#> Sum by sheets (axis =0):
#> array([[12, 14, 16, 18],
#> [20, 22, 24, 26],
#> [28, 30, 32, 34]])
#>
#> Sum by column-wise (axis = 1):
#> array([[12, 15, 18, 21],
#> [48, 51, 54, 57]])
#>
#> Sum by row-wise (axis = 2):
#> array([[ 6, 22, 38],
#> [54, 70, 86]])Heterogeneous data structure
Bei heterogenen Daten handelt sich um die Datenstrukturen bzw. Objekten, deren Elemente unterschiedlich sind. Z.B. in eine Liste können Objekte mit unterschiedlichen Datentypen gespeichert werden.
lists in R and lists & dicts in Python
lists in R
Unterschied zwischen der Liste und dem Vektor in R besteht darin, dass sie nicht homogen sein müssen und die Länge der Elemente unterschiedlich sein können. Zur Erstellung gibt es Funktion list() in R. Beim Indexieren werden die doppelte rechteckige Klammer [[]] benutzt (im Vergleich zum Vektor []).
# create a list
lst_r <- list(1, c(2, 3),
c(TRUE, FALSE),
c("I", "love", "R"), "Hello R")
#
lst_r
#> [[1]]
#> [1] 1
#>
#> [[2]]
#> [1] 2 3
#>
#> [[3]]
#> [1] TRUE FALSE
#>
#> [[4]]
#> [1] "I" "love" "R"
#>
#> [[5]]
#> [1] "Hello R"
cat("Type: ", class(lst_r), "\n")
#> Type: list
# Atribute of the list:
str(lst_r)
#> List of 5
#> $ : num 1
#> $ : num [1:2] 2 3
#> $ : logi [1:2] TRUE FALSE
#> $ : chr [1:3] "I" "love" "R"
#> $ : chr "Hello R"
#
# Check element (Hello R) is in the list
"Hello R" %in% lst_r
#> [1] TRUE
#
# Change the 1st element (1) to 42
lst_r[[1]] <- 42
lst_r[[1]]
#> [1] 42
#
# Remove the last element (Hello R) of the list
lst_r[[length(lst_r)]] <- NULL
lst_r
#> [[1]]
#> [1] 42
#>
#> [[2]]
#> [1] 2 3
#>
#> [[3]]
#> [1] TRUE FALSE
#>
#> [[4]]
#> [1] "I" "love" "R"
#
# Check element (Hello R) is in the list
"Hello R" %in% lst_r
#> [1] FALSE
#
# Remove the 1st (42) and 2nd elements ([2, 3])
(lst_r[-c(1,2)]) # Outer bracket for execute code direct
#> [[1]]
#> [1] TRUE FALSE
#>
#> [[2]]
#> [1] "I" "love" "R"Die Elemente von der Liste in R können auch benannt sein (wie in dictionaries in Python). Dabei kann man auf die Elemente durch [[]] oder durch den Operator $ (Dollar-Zeichen) zugreifen. In R kann man die Liste ändern oder entfernen, indem man dem Index von dem zu updatenden Element zu ändernde Wert zuweist.
lst_named <- list(
"age" = 19,
"sex" = "m",
"it_skills" = c("R", "Python", "Java")
)
#
# 1st element
lst_named[[1]]
#> [1] 19
#
# 3rd element
lst_named[[3]]
#> [1] "R" "Python" "Java"
#
# 2nd element of the 3rd element
lst_named[[3]][2]
#> [1] "Python"
#
# 2nd element by name
lst_named[["sex"]]
#> [1] "m"
#
# 3rd element via $
lst_named$it_skills
#> [1] "R" "Python" "Java"
#
# 2nd element from 3rd element from the list
lst_named$it_skills[2]
#> [1] "Python"
#
# Names of the list
names(lst_named)
#> [1] "age" "sex" "it_skills"Man kann in die Liste in R neues Element durch append() - Funktion hinzufügen oder sie in eine andere Datenstruktur wie Vektor oder Data Fareme (wenn sie ein named list ist) umwandeln.
# Add an element(\"Python\" and \"C++\") into the list(lst_r):
append(lst_r, c("Python", "C++"))
#> [[1]]
#> [1] 42
#>
#> [[2]]
#> [1] 2 3
#>
#> [[3]]
#> [1] TRUE FALSE
#>
#> [[4]]
#> [1] "I" "love" "R"
#>
#> [[5]]
#> [1] "Python"
#>
#> [[6]]
#> [1] "C++"
#
# Convert to vector
unlist(lst_r)
#> [1] "42" "2" "3" "TRUE" "FALSE" "I" "love" "R"
#
# Create alist
emp_list <- list(
emp_name = c("Max", "Ali", "Vali"),
emp_age = c(45, 34, 27),
emp_salary = c(5.300, 3.700, 3.300)
)
#
# Type of emp_list:
class(emp_list)
#> [1] "list"
#
# Convert to data frame
emp_df <- data.frame(emp_list); emp_df
#> emp_name emp_age emp_salary
#> 1 Max 45 5.3
#> 2 Ali 34 3.7
#> 3 Vali 27 3.3
#
# Type of emp_df
class(emp_df)
#> [1] "data.frame"lists in Python
Wie in R können auch die Elemente/Objekte in einer Liste in Python unterschiedliche Länge und Typen haben. Wenn alle Element der Liste in Python den gleichen Type haben, das ist diese vergleichbar mit dem Vektor in R.
# Create a list
lst_py = [1, [1, 2], True, ["I", "love", "Python"], "Hello Python"]
print(lst_py)
#> [1, [1, 2], True, ['I', 'love', 'Python'], 'Hello Python']
print("\nType: ", type(lst_py))
#>
#> Type: <class 'list'>
#
# Check element (Hello Python) is in the list
"Hello Python" in lst_py
#> True
#
# Change the 1st element (1) to 42
lst_py[0] = 42; lst_py
#> [42, [1, 2], True, ['I', 'love', 'Python'], 'Hello Python']
#
# Remove the last element (Hello Python) from the list
lst_py.pop(); lst_py # pop() removes the last element in the list!!
#> 'Hello Python'
#> [42, [1, 2], True, ['I', 'love', 'Python']]
#
# Remove the 2nd element ([1, 2]) with pop()
lst_py.pop(1); lst_py
#> [1, 2]
#> [42, True, ['I', 'love', 'Python']]
#
# Remove the 1st element (42) with del
del lst_py[-0]
#
# Remove by name
lst_py.remove(True)
lst_py
#> [['I', 'love', 'Python']]Neues Element in die Liste in Python kann durch append()- Funktion (wie in R) hinzugefügt werden. Die Listen kann man auch in Python wie in R in andere Datenstruktur umwandeln, in NumPy-Array, Pndas-Series oder Pandas-DataFrame.
# Create a list
ls1 = ["Ali", "Vali", "Gani"]; ls1
#> ['Ali', 'Vali', 'Gani']
#
# Add a new element (Max) into the list
# append() adds the new item to the end of the list
ls1.append("Max"); ls1
#> ['Ali', 'Vali', 'Gani', 'Max']
#
# insert() adds an element to the list at the specified index
ls1.insert(1, "Ivan") # inserted at index 1 (2nd position)
#
# Conver to a Pandas Series
ls_pd = pd.Series(ls1); ls_pd
#> 0 Ali
#> 1 Ivan
#> 2 Vali
#> 3 Gani
#> 4 Max
#> dtype: object
type(ls_pd)
#> <class 'pandas.core.series.Series'>
#
# Conver to a A Numpy 1d-Ayrray
ls_np = np.array(ls1); ls_np
#> array(['Ali', 'Ivan', 'Vali', 'Gani', 'Max'], dtype='<U4')
type(ls_np)
#> <class 'numpy.ndarray'>
#
# Convert to a Pandas DataFrame
ls2 = [3, 4, 5, 8, 2]
ls_df = pd.DataFrame(list(zip(ls1,ls2)), columns = ["ls1", "ls2"])
ls_df
#> ls1 ls2
#> 0 Ali 3
#> 1 Ivan 4
#> 2 Vali 5
#> 3 Gani 8
#> 4 Max 2dicts in Python and lists in R
Wie oben bereits erwähnt, in R müssen die Elemente einer Liste nicht vom gleichen Typ im Vergleich zu den Vektoren sein. Die Elemente in einer Liste in R können aber auch benannt sein, das nennt man dann named list. In Python dagegen können die Elemente nicht benannt sein, dafür aber gibt es in Python dictionanries, die gleiche Struktur wie named list in R haben. Dictionary in Python besteht aus d = {key : value} Struktur. Der Key bzw Schlüssel ist unique und immutable. Meistens werden für den Schlüssel strings benutzt. Die gleiche Struktur hat man in R, wenn man die Elemente in der Liste benennt: d = list(name = value). Zugriff auf einzelnes Element erfolgt durch d["key"] in Python und durch d["name"] oder d$name in R.
Folgendes Beispiel mit den Aktienkursen (Stand: 27.04.2024, von OnVista) möge es besser zum Ausdruck bringen.
show the code
share_name <- c("AMZN", "AAPL", "MBG", "TSLA", "BYD", "BABA", "SAP")
share_value <- c(179.62, 169.30, 74.36, 168.29, 25.36, 75.55, 174.00)
#
cat("Create a named list from vectors:\n")
dic_r <- setNames(as.list(share_value), share_name); dic_r
cat("\nshare price from BYD. slice with [[]]:\n")
dic_r[["BYD"]]
cat("\nshare price from Tesla. slice with $\n")
dic_r$TSLA
cat("\nTyp from dic_r: ", class(dic_r))
cat("\nAdd an element (BMW3) to the list(dic_r[['BMW3']] <- 99.15):\n")
dic_r[["BMW3"]] <- 99.15
cat("Is BMW3 there?\n")
dic_r["BMW3"] %in% dic_r
dic_r[["BMW3"]]
#
cat("\nRemove an element (MBG) from the list (dic_r$MBG <- NULL):\n")
dic_r$MBG <- NULL
cat("Is MBG there?\n")
dic_r["MBG"] %in% dic_r
# convert to python
cat("\nConvert the list (dic_r) to Python object (dic_py)\n")
dic_py <- r_to_py(dic_r)
dic_py
cat("\nType from dic_py:\n")
class(dic_py)#> Create a named list from vectors:
#> $AMZN
#> [1] 179.62
#>
#> $AAPL
#> [1] 169.3
#>
#> $MBG
#> [1] 74.36
#>
#> $TSLA
#> [1] 168.29
#>
#> $BYD
#> [1] 25.36
#>
#> $BABA
#> [1] 75.55
#>
#> $SAP
#> [1] 174
#>
#>
#> share price from BYD. slice with [[]]:
#> [1] 25.36
#>
#> share price from Tesla. slice with $
#> [1] 168.29
#>
#> Typ from dic_r: list
#> Add an element (BMW3) to the list(dic_r[['BMW3']] <- 99.15):
#> Is BMW3 there?
#> [1] TRUE
#> [1] 99.15
#>
#> Remove an element (MBG) from the list (dic_r$MBG <- NULL):
#> Is MBG there?
#> [1] FALSE
#>
#> Convert the list (dic_r) to Python object (dic_py)
#> {'AMZN': 179.62, 'AAPL': 169.3, 'TSLA': 168.29, 'BYD': 25.36, 'BABA': 75.55, 'SAP': 174.0, 'BMW3': 99.15}
#>
#> Type from dic_py:
#> [1] "python.builtin.dict" "python.builtin.object"show the code
print("share price at 27.04.2024:")
for k,v in r.dic_py.items():
print(f"{k} : {v}")
print("\nKeys:", r.dic_py.keys())
print("Values:", r.dic_py.values())
print(f"\nShare of Tesla: {r.dic_py["TSLA"]}")
print("\nAdd MBG to the dictionary(r.dic_py['MBG'] = 74.36):")
r.dic_py["MBG"] = 74.36
print("\nIs MBG in the dictionary?")
"MBG" in r.dic_py
print("\nDelete BYD from dictionary(del r.dic_py['BYD']):")
del r.dic_py["BYD"]
print("\nIs BYD in the dictionary?")
"BYD" in r.dic_py#> share price at 27.04.2024:
#> AMZN : 179.62
#> AAPL : 169.3
#> TSLA : 168.29
#> BYD : 25.36
#> BABA : 75.55
#> SAP : 174.0
#> BMW3 : 99.15
#>
#> Keys: dict_keys(['AMZN', 'AAPL', 'TSLA', 'BYD', 'BABA', 'SAP', 'BMW3'])
#> Values: dict_values([179.62, 169.3, 168.29, 25.36, 75.55, 174.0, 99.15])
#>
#> Share of Tesla: 168.29
#>
#> Add MBG to the dictionary(r.dic_py['MBG'] = 74.36):
#>
#> Is MBG in the dictionary?
#> True
#>
#> Delete BYD from dictionary(del r.dic_py['BYD']):
#>
#> Is BYD in the dictionary?
#> FalseZum Zugrif auf Elemente in einem Dictionary in Python gibt es neben dem Method dict["key"]noch das Method dict.get("key", "default"). Die beiden Methode liefern das gleiche Ergebnis. Unterschied besteht darin, ob der gesuchte Key in diectionary vorhanden ist oder nicht. Wenn Key nicht vorhanden ist, erhält man beim ersten Method den KeyError, das wird mit get() umgegangen. Standardmäsig wird None zurückgegeben, das kann man aber feststellen.
show the code
bsp_dic = {
"Ali" : 25,
"Max" : 44,
"Vali": 33
}; bsp_dic
print("With []:")
print(f"Alini yoshi: {bsp_dic["Ali"]}")
print("\nWith get():")
print(f"Alini yoshi: {bsp_dic.get("Ali")}")
lstName = ["Ali", "Gani", "Max", "Vali", "Brandon"]; lstName
print("\nIterate with devault value, if key not exis")
for n in lstName:
# for k, v in bsp_dic.items():
print(f"{n} - {bsp_dic.get(n, 'Mavjud emas!')}" )#> {'Ali': 25, 'Max': 44, 'Vali': 33}
#> With []:
#> Alini yoshi: 25
#>
#> With get():
#> Alini yoshi: 25
#> ['Ali', 'Gani', 'Max', 'Vali', 'Brandon']
#>
#> Iterate with devault value, if key not exis
#> Ali - 25
#> Gani - Mavjud emas!
#> Max - 44
#> Vali - 33
#> Brandon - Mavjud emas!sets and tuples in Python
In Python gibt es weitere Strukturen von Daten, set{} und tuple().
set{} in Python
Die gruppierten Elemente lassen sich in Python unter anderem auch durch set darstellen: die Elemente im geschweiften Klammern set{} eingeben oder in set()- Funktion übergeben. Besonderheit in diesem Konzept ist, dass die Elemente unique sind, d.h. ein Objekt kommt in Mengen nur einmal vor. In R gibt es die Funktion unique(), die doppelt vorkommende Elemente in einem Vektor entfernt. Desweiteren sind sie ungeordnet und besitzen keine Indizes, über die man auf Elemente zugreifen kann. Die Menge mit set - mutable, also veränderlich und mit frozenset -immutable, aslo nicht veränderbar.
Die Methoden in von sets in Python wie union, intersection oder difference gibt es auch in R, die man bei Vektoren anwenden kann.
# set() - returns the vector without duplicate values
my_lst = [2, 3, 1, 5, 3, 9, 3, 2, 7]; my_lst
#> [2, 3, 1, 5, 3, 9, 3, 2, 7]
my_set = set(my_lst); my_set
#> {1, 2, 3, 5, 7, 9}
#
# Create a set
x = {1, 2, 3, 4, 5, 6}; x
#> {1, 2, 3, 4, 5, 6}
y = set([4, 5, 6, 7, 8, 9]); y
#> {4, 5, 6, 7, 8, 9}
print(type(x), type(y))
#> <class 'set'> <class 'set'>
#
# union() - combine x and y without duplicates
x.union(y)
#> {1, 2, 3, 4, 5, 6, 7, 8, 9}
#
# intersection() - which obj in both sets
x.intersection(y)
#> {4, 5, 6}
#
# difference() - objects in x, but not in y
x.difference(y)
#> {1, 2, 3}
#
# difference() - objects in y, but not in x
y.difference(x)
#> {8, 9, 7}# Typ of python sets
class(py$x)
#> [1] "python.builtin.set" "python.builtin.object"
class(py$y)
#> [1] "python.builtin.set" "python.builtin.object"
#
# unique() - returns the vector without duplicate values
my_vec <- c(2, 3, 1, 5, 3, 9, 3, 2, 7); my_vec
#> [1] 2 3 1 5 3 9 3 2 7
uniq_vec <- unique(my_vec); uniq_vec
#> [1] 2 3 1 5 9 7
#
# create vectros
x_r <- c(1, 2, 3, 4, 5, 6); x_r
#> [1] 1 2 3 4 5 6
y_r <- c(4, 5, 6, 7, 8, 9); y_r
#> [1] 4 5 6 7 8 9
#
# union() - combine x_r and y_r without duplicates
union(x_r, y_r)
#> [1] 1 2 3 4 5 6 7 8 9
#
# intersection() - which obj in both vectors
intersect(x_r, y_r)
#> [1] 4 5 6
#
# setdiff() - objects in x_r, but not in y_r
setdiff(x_r, y_r)
#> [1] 1 2 3
#
# setdiff() - objects in y_r, but not in x_r
setdiff(y_r, x_r)
#> [1] 7 8 9tuple() in Python
Ein Tupel fasst mehrere (mit unterschiedlichen Typen) Elemente zu einem Objekt. Sie werden mit Komma getrennten Elementen in Klammern t = (obj1, obj2, ...), auch ohne Klammenr t = obj1, obj2, ... oder mit der Funktion tuple(obj) erzeugt. Im Vergleich zu den Listen sind Teuples immutable, man sagt auch unveränderbare Liste. Wenn man sie einmal erstellt, lassen sie sich nicht ändern, wenn das Ändern notwendig ist, kann man sie in die Liste umwandeln und nach der Änderung wieder zurück in den Tupel. Sie können auch in/mit Dictionaries verwendet werden.
# create a tuple
t1 = 1, 5, 6, 6, 3; print("t1: ", t1)
#> t1: (1, 5, 6, 6, 3)
t2 = (2, 4, 2, 5, 6); print("t2: ", t2)
#> t2: (2, 4, 2, 5, 6)
t3 = tuple([3, 4, 5, 6, 1]); print("t3: ", t3)
#> t3: (3, 4, 5, 6, 1)
#
# tuples in dict keys
koordinat = {
(0,0) : 100,
(1,1) : 200,
(0,1) : 75
}; koordinat
#> {(0, 0): 100, (1, 1): 200, (0, 1): 75}
# add istem
koordinat[(1,0)] = 125; koordinat
#> {(0, 0): 100, (1, 1): 200, (0, 1): 75, (1, 0): 125}
#
# tuples in dict values
greats = {
"Navoi" : ("Hirot", 1441),
"Bobur" : ("Andijon",1483),
"Temur" : ("Shahrisabz", 1336)
}; greats
#> {'Navoi': ('Hirot', 1441), 'Bobur': ('Andijon', 1483), 'Temur': ('Shahrisabz', 1336)}
#
# iterate dict
for great, (place, year) in greats.items():
print(f"{great} - was born in {place} in {year}.")
#> Navoi - was born in Hirot in 1441.
#> Bobur - was born in Andijon in 1483.
#> Temur - was born in Shahrisabz in 1336.data.frames in R and pandas.DataFrame in Python
Dataframes sind möchlicherweise die häufigste Art, die Daten in R und Python zu speichern, zu bearbeiten und zu analyisieren. Als DF (DataFrame) kann man sich eine Tabelle mit vier Ecken (Zeilen und Spalten) vorstellen, die gängigsten sind Excel Sheets. Die Zeilen stellen die Fälle oder Beobachtungen (engl. observations) und Spalten die Variable oder auch in der Statistik Merkmale (engl. futures) dar. Rechts ist Data Frame durch eine kleine Tabelle dargestellt. Die Tabelle hat 10 Zeilen (Beobachtungen = Namen) und 3 Spalten (Variable = Name, Geburtsjahr und Geburtsort).
| names | byear | bplace |
|---|---|---|
| Navoi | 1441 | Hirot |
| Bobur | 1483 | Andijon |
| Temur | 1336 | Shahrisabz |
| Cholpon | 1898 | Andijon |
| Qodiriy | 1894 | Toshkent |
| Ulugbek | 1394 | Samarqand |
| Xorazmi | 783 | Xiva |
| Beruniy | 1048 | Xorazm |
| Termiziy | 824 | Termiz |
| Buxoriy | 810 | Buxoro |
Data Frames sind in R praktisch eine benannte Liste von Vektoren (in Python wären pandas.Series) gleicher Länge, wobei jeder Vektor (Series) eine Spalte darstellt. Dataframes in R und Python besitzen einige Gemeinsamkeiten (Brown 2023), die dataframes voraussetzen.
- jede Spalte muss gleiche Länge haben
- alle Elemente in der Spalte müssen gleichen Typ haben
- Elemente in der Zeile können unterschiedlichen Type haben
- Zeilen und Spalten können unterschiedlich benannt werden
Sie werden in R mit data.frame() und in Python mit pandas pandas.DataFrame() erzeugt.
Create a DF
names <- c("Navoi", "Bobur", "Temur", "Cholpon", "Qodiriy",
"Ulugbek", "Xorazmi", "Beruniy", "Termiziy", "Buxoriy")
byear <- c(1441, 1483, 1336, 1898, 1894, 1394, 783, 1048, 824, 810)
bplace <- c("Hirot", "Andijon", "Shahrisabz", "Andijon", "Toshkent",
"Samarqand", "Xiva", "Xorazm", "Termiz", "Buxoro")
# Create a DataFrame -----------------------------------------
df <- data.frame(names, byear, bplace)
# Type -------------------------------------------------------
class(df)
#> [1] "data.frame"
# nums of rows and cols --------------------------------------
dim(df) # like shape in pandas
#> [1] 10 3
# num of rows ------------------------------------------------
nrow(df)
#> [1] 10
# num of columns ---------------------------------------------
ncol(df)
#> [1] 3
# structur of DataFrame --------------------------------------
# str(df) or
pillar::glimpse(df) # like pandas.DataFrame.info()
#> Rows: 10
#> Columns: 3
#> $ names <chr> "Navoi", "Bobur", "Temur", "Cholpon", "Qodiriy", "Ulugbek", "Xo…
#> $ byear <dbl> 1441, 1483, 1336, 1898, 1894, 1394, 783, 1048, 824, 810
#> $ bplace <chr> "Hirot", "Andijon", "Shahrisabz", "Andijon", "Toshkent", "Samar…
# First n lines, default = 6 ---------------------------------
head(df, n = 3)
#> names byear bplace
#> 1 Navoi 1441 Hirot
#> 2 Bobur 1483 Andijon
#> 3 Temur 1336 Shahrisabz
# Last n lines -----------------------------------------------
tail(df, n=2)
#> names byear bplace
#> 9 Termiziy 824 Termiz
#> 10 Buxoriy 810 Buxoro
# colnames ---------------------------------------------------
colnames(df)
#> [1] "names" "byear" "bplace"
# rows -------------------------------------------------------
rownames(df)
#> [1] "1" "2" "3" "4" "5" "6" "7" "8" "9" "10"# create pandas DataFrame from created df in r -------
df_pd = pd.DataFrame(r.df)
df_pd["byear"] = df_pd["byear"].astype(int)
# Type -----------------------------------------------
type(df_pd)
#> <class 'pandas.core.frame.DataFrame'>
# nums of rows and cols ------------------------------
df_pd.shape # like dim() in r
#> (10, 3)
# num of rows ----------------------------------------
len(df_pd)
#> 10
# num of columns -------------------------------------
len(df_pd.columns)
#> 3
# structur of DataFrame ------------------------------
df_pd.info() # like str() in r
#> <class 'pandas.core.frame.DataFrame'>
#> RangeIndex: 10 entries, 0 to 9
#> Data columns (total 3 columns):
#> # Column Non-Null Count Dtype
#> --- ------ -------------- -----
#> 0 names 10 non-null object
#> 1 byear 10 non-null int32
#> 2 bplace 10 non-null object
#> dtypes: int32(1), object(2)
#> memory usage: 332.0+ bytes
# First n lines, default = 5 -------------------------
df_pd.head(n = 3)
#> names byear bplace
#> 0 Navoi 1441 Hirot
#> 1 Bobur 1483 Andijon
#> 2 Temur 1336 Shahrisabz
# Last n lines ---------------------------------------
df_pd.tail(n = 2)
#> names byear bplace
#> 8 Termiziy 824 Termiz
#> 9 Buxoriy 810 Buxoro
# colnames -------------------------------------------
df_pd.columns
#> Index(['names', 'byear', 'bplace'], dtype='object')
# rows -----------------------------------------------
df_pd.index
#> RangeIndex(start=0, stop=10, step=1)Access a (the) element(s)
# 1st row, 1st col -------------------------------
df[1,1]
#> [1] "Navoi"
# 1-3 rows, all cols -----------------------------
df[1:3, ]
#> names byear bplace
#> 1 Navoi 1441 Hirot
#> 2 Bobur 1483 Andijon
#> 3 Temur 1336 Shahrisabz
# all rows, 1-2 cols -----------------------------
df[, 1:2]
#> names byear
#> 1 Navoi 1441
#> 2 Bobur 1483
#> 3 Temur 1336
#> 4 Cholpon 1898
#> 5 Qodiriy 1894
#> 6 Ulugbek 1394
#> 7 Xorazmi 783
#> 8 Beruniy 1048
#> 9 Termiziy 824
#> 10 Buxoriy 810
# 3 rows, col bplace -----------------------------
df[1:3, 3]
#> [1] "Hirot" "Andijon" "Shahrisabz"
# rows where col bplace = "Andijon" --------------
df[df$bplace == "Andijon", ]
#> names byear bplace
#> 2 Bobur 1483 Andijon
#> 4 Cholpon 1898 Andijon
# rows where col byear < 1000 -------------------
df[df$byear < 1000, ]
#> names byear bplace
#> 7 Xorazmi 783 Xiva
#> 9 Termiziy 824 Termiz
#> 10 Buxoriy 810 Buxoro# 1st row, 1st col -------------------------------------
df_pd.iloc[0,0] # iloc by postion(s)
#> 'Navoi'
# 1-3 rows, all cols -----------------------------------
df_pd.iloc[:3, ]
#> names byear bplace
#> 0 Navoi 1441 Hirot
#> 1 Bobur 1483 Andijon
#> 2 Temur 1336 Shahrisabz
# all rows, 1-2 cols -----------------------------------
df_pd.iloc[:, :2]
#> names byear
#> 0 Navoi 1441
#> 1 Bobur 1483
#> 2 Temur 1336
#> 3 Cholpon 1898
#> 4 Qodiriy 1894
#> 5 Ulugbek 1394
#> 6 Xorazmi 783
#> 7 Beruniy 1048
#> 8 Termiziy 824
#> 9 Buxoriy 810
# 3 rows, col bplace -----------------------------------
df_pd.loc[:3, "bplace"] # loc by labels(s)
#> 0 Hirot
#> 1 Andijon
#> 2 Shahrisabz
#> 3 Andijon
#> Name: bplace, dtype: object
# 1-2 rows, cols names and bplace ----------------------
df_pd.loc[:2, ["names","byear"]] # or df_pd.bplace
#> names byear
#> 0 Navoi 1441
#> 1 Bobur 1483
#> 2 Temur 1336
# rows where col bplace = "Andijon" --------------------
df_pd.loc[df_pd["bplace"] == "Andijon"]
#> names byear bplace
#> 1 Bobur 1483 Andijon
#> 3 Cholpon 1898 Andijon
# rows where col byear < 1000 --------------------------
df_pd.loc[df_pd.byear < 1000]
#> names byear bplace
#> 6 Xorazmi 783 Xiva
#> 8 Termiziy 824 Termiz
#> 9 Buxoriy 810 Buxoro
# ------------------------------------------------------Change Row- and Colnames
show the code
cat("Rownames:\n", rownames(df)) # rownames
cat("\nColnames:", colnames(df)) # colnames
#
new_rname <- paste("person",1:10, sep="_", collapse= ",")
new_rname <- unlist(strsplit(new_rname, ","))
cat("\n\nNew Rowsname:\n", new_rname)
cat("\nDF with modified rownames:\n")
# change the rownames
rownames(df) <- (new_rname)
head(df, n = 2)
# change the colnames
# Method1: colnames(c(names of new columns))
# Method2: setNames(df, c(names of new columns))
# Here will be used Method2
# change colname
df <- setNames(df, c("Ism", "Tug\'ilgan yil", "Tug\'ilgan joy"))
cat("\n\nDF with modified colnames:\n")
head(df, n = 2)
#
cat("\n\nSet key in DF with data.table:\n")
dt <- data.table(df)
setkey(dt, Ism) # Like Index in pandas
#call("Key: ", key(dt))
head(dt, n = 2)#> Rownames:
#> 1 2 3 4 5 6 7 8 9 10
#> Colnames: names byear bplace
#>
#> New Rowsname:
#> person_1 person_2 person_3 person_4 person_5 person_6 person_7 person_8 person_9 person_10
#> DF with modified rownames:
#> names byear bplace
#> person_1 Navoi 1441 Hirot
#> person_2 Bobur 1483 Andijon
#>
#>
#> DF with modified colnames:
#> Ism Tug'ilgan yil Tug'ilgan joy
#> person_1 Navoi 1441 Hirot
#> person_2 Bobur 1483 Andijon
#>
#>
#> Set key in DF with data.table:
#> Key: <Ism>
#> Ism Tug'ilgan yil Tug'ilgan joy
#> <char> <num> <char>
#> 1: Beruniy 1048 Xorazm
#> 2: Bobur 1483 Andijonshow the code
print(f"Rownames(index):\n{list(df_pd.index)}")
print(f"\nColnames: {list(df_pd.columns)}")
#
new_pname = ["user_" + str(idx) for idx in list(df_pd.index)]
print(f"\nNew Rowsname:\n{new_pname}")
print("\nDF with modified rownames:")
df_pd.index = new_pname
df_pd.head(n = 2)
print("\nDF with modified colnames:")
df_pd.columns = ["Ism", "Tug\'ilgan yil", "Tug\'ilgan joy"]
df_pd.head(n = 2)
print("\n\nSet a column as index:")
# like key in data.table in r
df_pd.set_index("Ism", inplace = True, append = True)
df_pd.head(n = 2)#> Rownames(index):
#> [0, 1, 2, 3, 4, 5, 6, 7, 8, 9]
#>
#> Colnames: ['names', 'byear', 'bplace']
#>
#> New Rowsname:
#> ['user_0', 'user_1', 'user_2', 'user_3', 'user_4', 'user_5', 'user_6', 'user_7', 'user_8', 'user_9']
#>
#> DF with modified rownames:
#> names byear bplace
#> user_0 Navoi 1441 Hirot
#> user_1 Bobur 1483 Andijon
#>
#> DF with modified colnames:
#> Ism Tug'ilgan yil Tug'ilgan joy
#> user_0 Navoi 1441 Hirot
#> user_1 Bobur 1483 Andijon
#>
#>
#> Set a column as index:
#> Tug'ilgan yil Tug'ilgan joy
#> Ism
#> user_0 Navoi 1441 Hirot
#> user_1 Bobur 1483 AndijonData Frames, wie schon erwähnt, wahrscheinlich die meist benutzten Datenstrukturen sowohl in R als auch in Python. Daher wurde das Thema hier kurz dargestellt und Fokus nur auf Basisfunktionen gelegt. Dazu wird es ein eigenes Thema geben.